En identifiant les types de cellules responsables de cette allergie alimentaire douloureuse, l’œsophagite à éosinophiles (EoE), cette équipe du Cincinnati Children's Hospital (Ohio) atteint plusieurs objectifs. Elle développe une plateforme capable de séquencer l'ARN monocellulaire qui sera précieuse à la recherche portant sur les différents types d’allergies. Elle identifie précisément les cellules T et les gènes responsables de l’EoE, apportant ainsi l’espoir de traitements pour cette condition handicapante. Enfin, elle soulève également des questions sur un complément alimentaire populaire, le butyrate, aux effets plutôt bénéfiques pour le système digestif inférieur mais plutôt néfastes dans l’œsophage…
L’incidence de l’œsophagite à éosinophiles (EoE), une maladie immunitaire chronique, qui provoque une inflammation de l'œsophage avec des symptômes de RGO, de dysphagie et d’impaction alimentaire (blocage des aliments dans l’œsophage) est en forte augmentation. La condition touche environ une personne sur 2.000. Cette maladie allergique chronique est caractérisée par un excès d’éosinophiles (un type de globule blanc) qui provoquent une inflammation rendant la déglutition et l’alimentation douloureuses. Certains patients finissent par être contraints de se nourrir avec des formules liquides.
L’aboutissement d’une recherche, initiée il y a plus de 8 ans, sur les cellules à l'origine de l’EoE, a fait du Cincinnati Children's le principal centre américain de traitement et d'étude de la maladie. Les chercheurs ont trouvé un nouveau moyen de traiter la maladie. Mais ils soulèvent aussi des questions sur un complément alimentaire souvent pris pour réduire l'inflammation intestinale.
L’équipe a développé et perfectionné un processus de 10 à 12 heures qui isole et analyse des cellules vivantes individuelles à partir d'échantillons de tissus humains de la taille de trois graines de sésame et s'est concentrée sur les cellules immunitaires à mémoire spécifique dans les tissus, qui répondent directement aux allergènes alimentaires. Cette analyse a également permis d'identifier les propriétés jusque-là inconnues de ces cellules. Parmi les principales observations et conclusions :
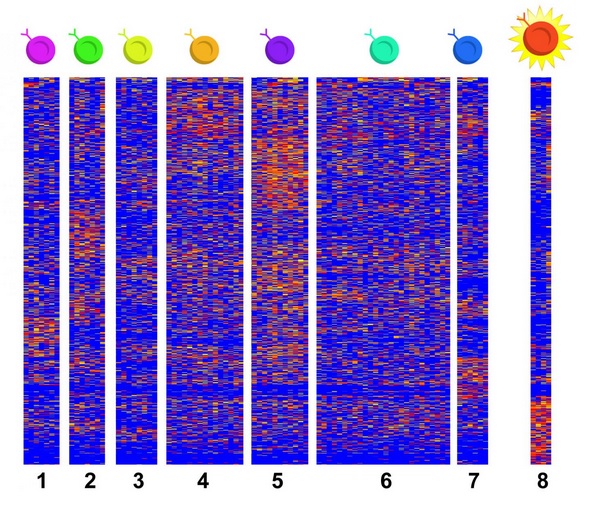
- 8 types de cellules T (du système immunitaire) sont identifiés dans le tissu œsophagien. C'est plus qu’on ne le pensait auparavant ;
- 2 types de cellules, T7 et T8, apparaissent en plus grand nombre dans le tissu malade ;
- l'un de ces 2 types de cellules réagit aux allergènes en produisant des quantités énormes de cytokines de type 2 responsables de l'inflammation ;
- l'un de ces types de cellules (T8) est presque inexistant dans les tissus sains, ce qui en fait une cible particulièrement intéressante pour le traitement : « l'inhibition de ces cellules est maintenant un objectif majeur de nos recherches », écrivent les chercheurs ;
- l'un des gènes hyperactifs qui contribue à déclencher la réponse inflammatoire (FFAR3) code également pour un récepteur d'un complément alimentaire courant, le butyrate. Le butyrate est un acide gras à chaîne courte (vendu comme supplément en ligne et dans de nombreux magasins, sans ordonnance). Dans le côlon, cet acide gras aide le corps à produire de l'énergie à partir de fibres alimentaires. Il semble également prévenir ou réduire l'inflammation dans le système digestif inférieur, ce qui l'a rendu populaire auprès des personnes atteintes de MICI. Cependant, dans l'œsophage, cet acide gras peut entraîner ou aggraver l'EoE en activant la population identifiée de cellules T productrices de cytokines. D'autres études seront nécessaires pour déterminer si patients atteints d'EoE aggravent leur condition en prenant du butyrate et si la prise de butyrate pour un problème du tube digestif inférieur augmente le risque de développer une éruption cutanée…
Cette première étude à utiliser le séquençage d'ARN monocellulaire pour recueillir des données sur les cellules sprésentes dans des tissus affectés par l'EoE a permis la réalisation d'une carte thermique (Visuel) qui présente les données de ces 8 types de cellules T. Au-delà, le processus utilisé pourrait être appliqué à d'autres conditions plus courantes pour identifier de petits groupes de cellules « coupables » et cachées dans les tissus.
Ainsi, la plateforme pourrait être utile dans l'étude de l'asthme, de la dermatite atopique, de la rhinite allergique et d'autres maladies associées aux cytokines de type 2.
Source: Journal of Clinical Investigation April 8, 2019 DOI : 10.1172/JCI125917 Single-cell RNA sequencing identifies inflammatory tissue T cells in eosinophilic esophagitis (Visuel Cincinnati Children's)
Plus sur Allergies Blog

Laisser un commentaire